Molekulare Struktur der DNA
Contributor(s)
| Written | 2006-10-01 | Jean-Loup Huret |
| Genetics |
Abstract
Molekulare Struktur der DNAContent
MolekulareStruktur der DNA
*
I Primärstruktur des Moleküls: kovalentesRückgrad und BasenI-1 PhosphorsäureI-2 ZuckerI-3 Stickstoffhaltige BasenII Senkundär- undTertiärstruktur des Moleküls – 3-dimensionaleKonformation der DNAII.1 DinukleotideII.2 DNA MolekülII.2.1 Wasserstoffbrücken: BasenpaarungII.2.2 Groβe Furche und kleine FurcheII.3 Non-B DNAII.3.1 Z-DNAII.3.2 kreuzförmige DNA-struktur und Haarnadel-DNAII.3.3 H-DNA oder Triplex-DNAII.3.4 G4-DNAIII Quartärstruktur desMoleküls - ChromatinIV VerschiedenesIV.1 DNA und MitochondrienIV.2 DNA Denaturierung |
*
Desoxyribonukleinsäure(DNA) ist die genetische Information der meistenlebenden Organismen (im Gegensatz dazu verwenden einige Viren,genannt Retroviren, Ribonukleinsäure als genetischeInformation).
- DNA kann über Generationen von Zellenkopiert werden: DNA Replikation,
- DNA kann in Proteinetranslatiert werden: DNA Transkription in RNA, weiter translatiert inProteine,
- DNA kann, wenn notwendig, repariert werden:DNA-Reparatur-Mechanismen ,
Ribonukleinsäuren (RNAs) werdenin einem anderen Kapitel beschrieben (mRNA, r-RNA, t-RNA...)
-DNA ist ein Polymer, das aus Einheiten aufgebaut ist, die Nukleotide(oder Mononukleotide) genannt werden.
- Nukleotide haben auchandere Funktionen: Energieträger (ATP, GTP), zelluläreAtmung (NAD, FAD); Signaltransduktion (zyklisches AMP); Koenzyme(CoA, UDP); Vitamine (Nicotinamid-Mononukleotid, Vit B2).
DerProteinnomenklatur folgenden können wir von Primär-,Sekundär-, Tertiär- und Quartärstruktur des Molekülssprechen:
I Primärstrukturdes Moleküls: kovalentes Rückgrad und Basen
EinNukleosid besteht aus einem Zucker plus einer stickstoffhältigenBase.
Ein Nukleotid besteht aus einem Phosphat+ einem Zucker+einer stickstoffhaltigen Base. In der DNA ist das Nukleotid einDesoxyribonukleotid (in der RNA ein Ribonukleotid).
I-1Phosphorsäure
Gibteine Phosphatgruppe.

Fig. 1
I-2Zucker
Desoxyriboseist eine zyklische Pentose (Zucker mit 5 Kohlenstoffen). Beachte: inder RNA ist der Zucker eine Ribose.
Kohlenstoffeim Zucher werden 1’ bis 5’ benannt. Um ein Nukleotid zubilden, ist ein Stickstoffatom der stickstoffhaltigen Base an das C1’gebunden (glykosidische Bindung), das Phosphat an das C5’(Esterbindung). Das Nukleotid ist daher: Phosphat – C5’Zucker C1’ – Base.

Fig. 2
I-3Stickstoffhaltige Basen
StickstoffhaltigeBasen sind aromatische Heterozyklen; es gibt Purine und Pyrimidine.
-Purine: Adenin (A) und Guanin (G).
- Pyrimidine: Cytosin (C) undThymin (T) (Beachte: Thymin wird in der RNA durch Uracyl (U)ersetzt).
Anmerkung:es gibt andere stickstoffhaltige Basen, insbesondere methylierteBasen, die von den oben genannten abstammen; Methylierung einige derBasen haben funktionelle Rolle (siehe dieses Kapitel).
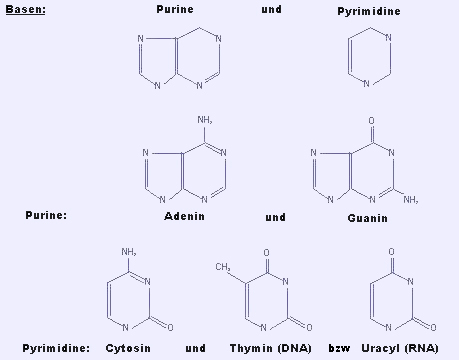
Fig. 3
Glossar:
-Nukleosidnamen: Desoxyribonukleoside in der DNA: Desoxyadenosin,Desoxyguanosin, Desoxycytidin, Desoxythymidin (Ribonukleoside in derRNA: Adenosin, Guanosin, Cytidin, Uridin).
- Nukleotidnamen:Desoxyribonukleotide in der DNA: Desoxyadenylsäure,Desoxyguanidylsäure, Desoxycytidylsäure,Desoxythymidylsäure (Ribonukleotide der RNA: Adenosinsäure,Guanylsäure, Cytidinsäure, Uridylsäure).

Fig. 4
IISekundär- und Tertiärstruktur des Moleküls –3-dimensionale Konformation der DNA
II.1Dinukleotide
Dinukleotideentstehen durch eine Phosphodiesterbindung zwischen 2Mononukleotiden. Das Phosphat eines Mononukleotids (am C5’seines Zuckers) wird an das C3’ des Zuckers des vorhergehendenMononukleotids gebunden. Somit ist, wenn wir mit einem Phosphatbeginnen, ein 5’ Zucker (+ Base) über das 3’ diesesZuckers mit dem 2. Phosphat und damit mit dem 5’ desdarauffolgenden Zuckers verbunden, dessen 3’ wiederum frei fürden nächsten Schritt ist. Die Bindung bzw die Orientierung desMoleküls ist also 5’->3’.
Polynukleotidewerden aus der sukzessiven Addition von Monomeren in einerallgemeinen 5’->3’ Konfiguration gebildet. DasRückgrad des Moleküls wird durch eine Abfolge vonPhosphat-Zucker (Nuckleotid N) – Phosphat-Zucker (NukleotidN+1) usw gebildet, die Bindungen sind kovalent, mit der Base seitlichdes Phosphat-Zuckers.

Fig. 5
II.2DNA Molekül
DNAbesteht aus 2 ("Duplex DNA") dextrogyre (wie eine Schraube,rechtsgängig) Helixketten oder –stränge ("dieDoppelhelix "), die um eine Achse mit einem Durchmesser von 20gedreht ist.
Die beiden Stränge sind antiparallel (d.h.ihre 5’->3’ Orientieren geht in entgegengesetzteRichtungen). Das Aussehen eines DNA-Polymers zeigt eine Periodizitätvon 3.4 , was einem Abstand zwischen 2 Basen entspricht, bzw von 34 ,was einer Umdrehung der Helix entspricht (also 10 Basenpaaren).

Fig. 6
II.2.1Wasserstoffbrücken: Basenpaarung
Die(hydrophoben) Basen sind innen aufeinander gestapelt, diese Ebenensind normal (90- Winkel) zur Achse der Doppelhelix. Die Auβenseite(Phosphat-Zucker) ist hydrophil.
Wasserstoffbrückenzwischen den Basen des einen und denen des zweiten Stranges haltendie beiden Stränge zusammen (gestrichelte Linie in derZeichnung).
EinPurin eines Stranges ist mit einem Pyrimidin auf dem 2. Strangverbunden. Folglich entspricht die Anzahl der Purinmonomere derAnzahl der Pyrimidinmonomere.
A bindet T (mit 2Wasserstoffbrücken).
G bindet C (mit 3 Wasserstoffbrücken;dies ist eine stabilere Bindung: 5.5 vs 3.5 kcal).
Anmerkung:die Anzahl der As entspricht der der Ts, und die Anzahl der Gsentspricht der der Cs in der DNA.
Die strikte Übereinstimmung(A<->T und G<->C) macht die beiden Stängekomplementär. Ein Strang ist die Matrize für den zweitenund umgekehrt: diese Eigenschaft erlaubt exakte Replikation(semi-konservative Replikation: ein Strang – die Matrize –ist konserviert, der zweite wird neu synthetisiert, dasselbe gibt fürden 2. Strang, der erhalten bleibt, was erlaubt, daβ der 1. neusynthetisiert wird. Siehe angrenzendes Kapitel.

Fig. 8
Anmerkung:
Wasserstoffbrückenin der Basenpaarung weichen manchmal vom Watson & Crick Modellab, das oben beschrieben ist, indem das N7 Atom eines Purinsverwendet wird anstelle des N1 (Hoogsteen Modell).

Fig. 9

Fig. 10
II.2.2Groβe Furche und kleine Furche
DieDoppelhelix ist ein relativ steifes und viskoses Molekül vonunglaublicher Länge und mit kleinem Durchmessers. Es enthälteine groβe und eine kleine Furche. Die groβe Furch ist tiefund weit, die kleine ist eng und flach.
DNA-Protein-Interaktionensind essentielle Prozesse im Leben einer Zelle(Transkriptionsaktivation oder -repression, DNA-Replikation und-Reparatur).
Proteine binden am Grund der Furchen, indem siespezifische Bindungen nutzen: Wasserstoffbrücken, oder auchnicht-spezifische Bindung: van der Waals Interaktionen, allgemeineelektrostatische Interaktionen.
Proteine erkennenWasserstoffdonoren und -akzeptoren, Methylgruppen (hydrophob),letztere sind ausschlieβlich in der groβen Furche; es gbt 4mögliche Erkennungsmuster für die groβe, aber nur 2für die kleine Furche (siehe Figur).
Einige Proteinebinden DNA an/in ihrer groβen Furche, andere an/in der kleinenFurche, einige müssen an/in beide/n binden.
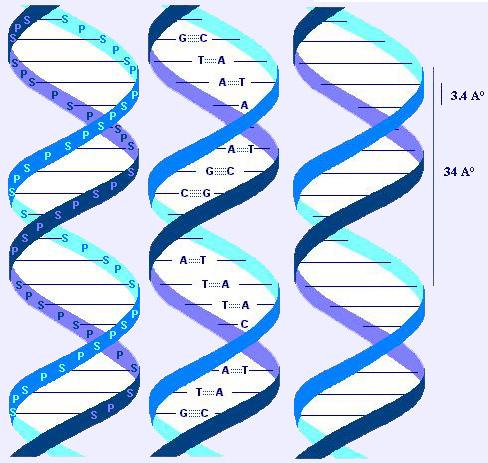
Fig. 11
Anmerkung:
-Die beiden Stänge werden "plus" und "minus"Stränge genannt, oder "direkt" und "revers".An einer gegebenen Stelle, wo ein Strang (einer der beiden) einekodierende Sequenz trägt, ist es unwahrscheinlich (aber nichtunmöglich), daβ der zweite Strang ebenfalls eine kodierendeSequenz enthält.
- DNA wird in vivo ionisiert und verhältsich wie ein Polyanion.
DieDoppelhelix wie oben beschrieben ist die "B"-Form der DNA;es ist die häufigste Form der DNA in vivo, es existieren aberandere Formen in vivo (siehe unten) oder in vitro. Die "A"-Formentspricht der B-DNA aber ist weniger hydriert als B-DNA, die"A"-Form kommt in vivo nicht vor.
II.3Non-B DNA
DNAist ein Molekül, das sich bewegt, zappelt, Gymnastik betreibt,tanzt. Die unten beschriebenen Strukturen haben bewiesenermaβenfunktionelle Rolle; andererseits scheinen sie DNA-Brüche,Deletionen, Amplifikationen, Rekombination und Mutationen zubegünstigen.
Glossar:
Palindrome:das sind Wörter oder Sätze, die von vorne und von hintengelesen dasselbe ergeben (z.B. "DNA LAND"). DNA spieltgerne mit Palindromen (siehe unten).
II.3.1Z-DNA
-Die Z-Form ist eine levogyre (linksgängige) Doppelhelix miteiner zick-zack-Konformation des Rückgrads (weniger glatt alsB-DNA). Nur eine Furche ist vorhanden, die der kleinen Furche ähnelt,die Basenpaare (die in der B-DNA die groβe Furche bilden, nahean der Achse) sind seitlich abgesetzt an der äuβerenOberfläche weit weg von der Achse. Phosphate sind näherbeieinander als in B-DNA. Z-DNA kann keine Nukleosomen bilden. -Hoher GC-Gehalt fördert Z-Konformation. Cytosin-methylierung undMoleküle, die in vivo vorhanden sind (wie z.B. Spermin undSpermidin) können Z-Konformation stabilisieren.
-DNA-Sequenzen können von der B-Form in die Z-Form springen undzurück: Z-DNA ist in vivo eine vorübergehenden Form.
-Z-DNA-Bildung geschieht während der Transkription von Genen anden Transkriptionsstartstellen nahe der Promotoren eines aktivtranskribierten Gens. Während der Transkription induziert dieBewegung der RNA-Polymerase ein negatives Supercoiling oberhalb undein positives Supercoiling unterhalb der Transkriptionsstelle. Dasnegative Supercoiling oberhalb fördert Z-DNA Bildung; eine derZ-DNA Funktionen könnte sein, das negative Supercoilong zuabsorbieren. Am Ende der Transkription bringt Topoisomerase die DNAzurück in die B-Konformation.
- Bestimmte Proteine bindenan Z-DNA, insbesondere doppelstängige RNA Adenosindeaminase(ADAR1), ein Z-DNA bindendes Enzym, das nukleare RNA editiert; diesesEnzym konvertiert Adenin zu Inosin in der prä-mRNA. Daraufhininterpretieren die Ribosomen Inosin als Guanin, wodurch sich daskodierte Protein mit seiner epigenetischen Modifikation verändert(siehe Kapitel Epigenetik).
Anmerkung:
-Z-DNA Antikörper gibt es in Lupus erythematosus und anderenAutoimmunerkrankungen.
- Doppelsträngige RNA (dsRNA) kannebenfalls Z-Konformation annehmen.
II.3.2Kreuzförmige DNA-struktur und Haarnadel-DNA
-Holliday Junctions (während der Rekombination gebildet) sindkreuzförmige Stukturen. Invertierte (oder Spiegel-)Wiederholungen (Palindrome) von Polypurin /Polypyrimidin-DNA-Regionen können durch intra-Strangpaarungebenfalls kreuzförmige oder Haarnadelstrukturen annehmen.
-Palindromische AT-reiche Wiederholungen findet man an denBruchpunkten von t(11;22)(q23;q11), der einzig bekannten immer wiedervorkommenden, angeborenen rezibroken Translokation.
- Nukleasenbinden und schneiden Holliday-Junctions nach der Rekombination.Andere gut bekannte Proteine wie die HMG Proteine oder MLL(weiterführend siehe: MLL)können ebenfalls kreuzförmige DNA binden.

Fig. 12

Fig. 13
II.3.3H-DNA oder Triplex-DNA
-Invertierte Wiederholungen (Palindrome) von Polypurin- bzwPolypyrimidin-DNA-Regionen können Triplexstrukturen bilden(Tripel–Helix). Ein Tripel-Strang plus ein einzelner Strangwerden dabei gebildet.
- H-DNA könnte eine Rolle alsfunktioneller Regulator bei der Genexpression haben oder auch aufRNAs wirken (z.B. Repression der Transkription).

Fig. 14
II.3.4G4-DNA
-G4-DNA oder Quadruplex-DNA: die Faltung einer doppelsträngigerGC-reichen Sequenz auf sich selbst, wodurch eine HoogsteenBasenpaarung zwischen 4 Guaninen ("G4"), eine hoch-stabileStuktur, gebildet wird. Oft wird sie nahe Promotoren von Genen und anden Telomeren gefunden.
- Rolle in Meiose und Rekombination;könnte ein regulatorische Element darstellen.
-RecQ-Familie der Helikasen ist fähig, G4-DNA zu entwinden (z.B.BLM, ein Gen, das im Blooms Syndrom mutiert ist; weiterführendesiehe: Bloomsyndrome).
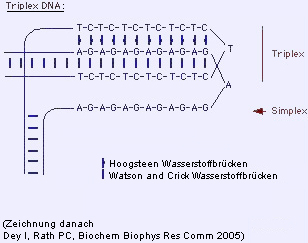
Fig. 15

Fig. 16
IIIQuartärstruktur des Moleküls - Chromatin
DNAist mit Proteinen assoziiert: Histone und nicht-Histonproteine,wodurch Chromatin gebildet wird. DNA als ganzes ist sauer (negativgeladen) und bindet basische Proteine (positiv geladen) genanntHistone: siehe Kapitel Chromatin .
Esgibt 3 x 10 9 Nukleotidpaareim menschlichen haploiden Genom, was ungefähr 30.000 Genenenspricht, die über 23 Chromosomen eines haploidenChromosomensatzes verteilt sind.
IVVerschiedenes
IV.1DNA und Mitochondrien
Sieheauch: Mitochondrialinheritance
-DNA findet sich im Nukleus einer Zelle, aber eine kleine Menge gibtes auch in den Mitochondrien.
- Mitochondrien stammen vonArchaebakterien ab, die Endosymbionten in einer eukaryotischen Zellewurden.
- Der genetische Code ist verschieden vom sogenannten"Universalcode" (UGA, AUA, AGA, AGG: STOP, Ile, Arg, ARG imUniversalcode; Trp, Met, STOP, STOP in den Mitochondrien von Säugernbzw andere Bedeutung in den Mitochondrien anderer Arten).
- DieAnzahl der DNA-Kopien in einem gegebenen Mitochondrium istvariabel.
- Mitochondriale DNA ist zirkulär, hat schwereund leichte Ketten, hat keine Introns und keine nicht-kodierendenSequenzen.
- Mitochondriale Gene kodieren für Proteine, dieim Elektronentransport, in ribosomalen RNAs (rRNAs) und transfer RNAs(tRNAs) eine Rolle spielen.
- Jeder DNA-Strang wirdtranskribiert, danach in mRNAs geschnitten, aber auch in rRNAs undtRNAs.
Anmerkung:Mitochondrien verwenden auch Proteine, die aus dem Zytoplasma derZelle importiert und vom Nukleus kodiert werden; bis jetzt wurdennoch keine Proteine gefunden, die von den Mitochondrien insZytoplasma exportiert werden, ausgenommen Apoptose.
IV.2DNA Denaturierung
DieDoppelhelix wird in vitro bei Erwärmung entwunden, bei extremenpH Werten und unter anderen Bedingungen (Harnstoff, ...). EinSchmelzpunkt kann errechnet werden; er ist charakterisiert duch dieA/T vs G/C Proportion des studierten Organismus, und zwar dadurch,daβ es nur 2 Wasserstoffbrücken bei A/T aber 3 bei G/Cgibt, letztere stellt eine stabilere Bindung dar. Bei Denaturierungverändern sich die physikalischen Eigenschaften der DNA; z.B.kommt es zu einem hyperchromischen Effekt: Lichtabsorption bei 260 nmist höher bei denaturierter DNA als bei doppelsträngigerDNA. Lichtabsorption variiert auch bzgl des A/T vx G/C Gehalts: erist höher in A/T-reichen als in G/C-reichenOrganismen.
DNA-Denaturierung sollte man kennen, da sie:
1-die Messung des A/T- vs G/C-Gehalts erlaubt;
2- die Basis vonHybridisierungstechnikek ist (in situ Hybridisierung, blots;Siehe Methodsin Genetics).
Ubersetzung: Katrina Vanura
Contributor(s) |
Written | 2006-10 | Jean-Loup Huret |
Genetics,Dept Medical Information, University of Poitiers, CHU PoitiersHospital, F-86021 Poitiers, France |
Keywords
Molekulare Struktur der DNACitation
Jean-Loup Huret
Atlas of Genetics and Cytogenetics in Oncology and Haematology 2022-05-17
Molekulare Struktur der DNA
Online version: http://atlasgeneticsoncology.org/teaching/209048/molekulare-struktur-der-dna
